Methodenentwicklung in der Bioinformatik
Methodenentwicklung im Kompetenzbereich Bioinformatik
Aus unserem Forschungsansatz der molekular erklärbaren Intelligenz sind mehrere Projekte zu methodischen Entwicklungen hervorgegangen. Diese reichen von Deep Learning Ansätzen zur präzisen Lokalisierung von Krankheitsmustern hin zur geometrischen Optimierung von für die Laser-Capture Mikrodissektion. Auch Software-Plattformen für die Infrarot-Mikroskopie gehören zu unseren methodischen Entwicklungen.
Deep Learning Ansätze zur präzisen Lokalisierung von Krankheitsmustern
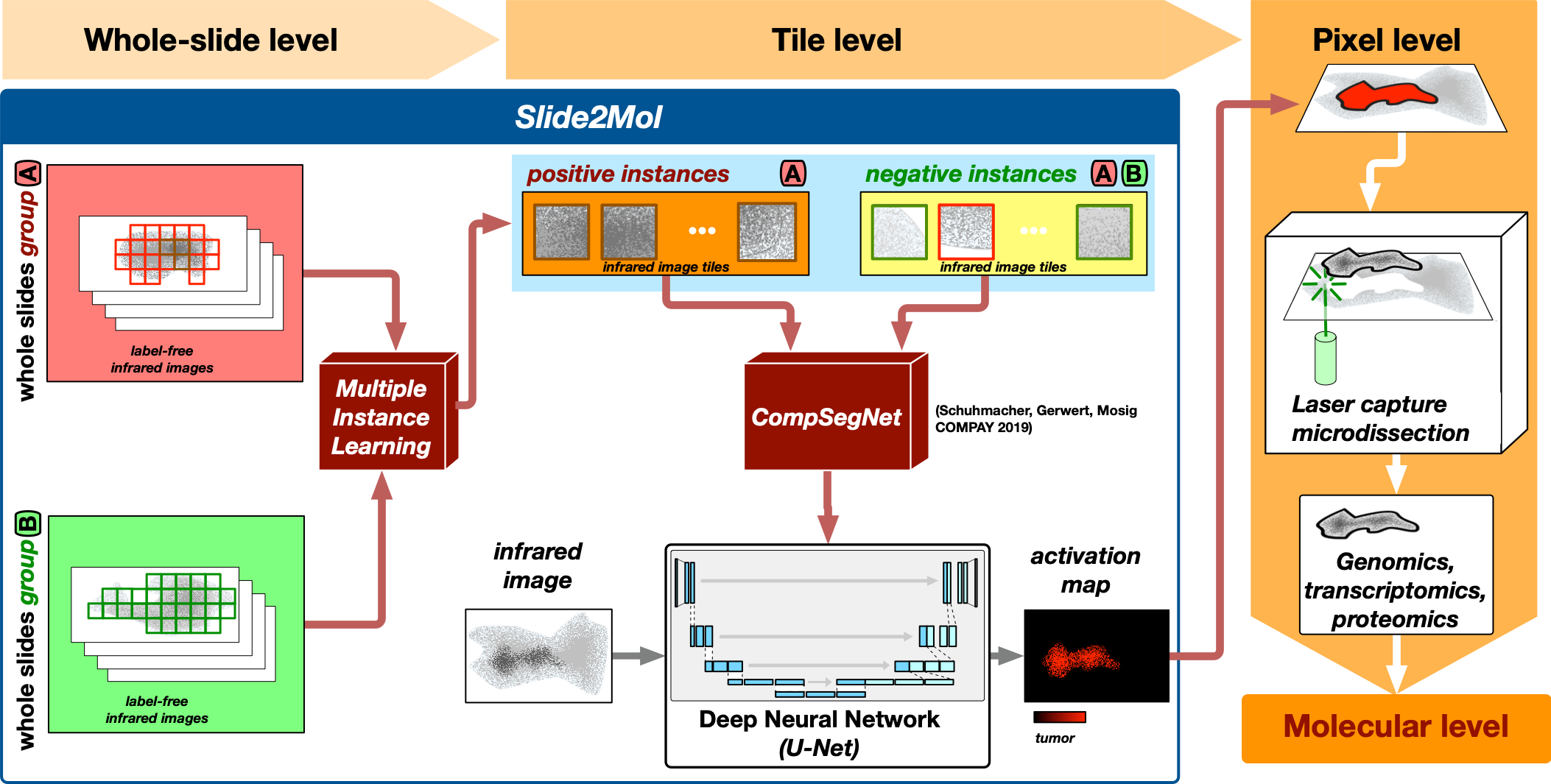
Mikroskopische Aufnahmen von Gewebedünschnitten gehen mit enormen Datenmengen einher. Infrarot-Mikroskopische Bilddaten von Gewebeproben umfassen hunderte von Millionen von Infrarot-Spektren, die jeweils einen ortsspezifischen molekularen Fingerabdruck der Probe liefern. In der Bioinformatik beschäfigen wir uns mit dem Problem, aus diesen enormen Mengen ortsspezifischer Spektralinformation die krankheitsspezifischen Regionen zu lokalisieren. Hierzu verwenden wir verschiedene Deep-Learning Ansätze, neben dem Multiple Instance Learning auch andere Ansätze des sogenannten schwach überwachten Lernens, beispielsweise das Comparative Segmentation Network.
Geometrische Optimierung für die Laser-Capture Mikrodissektion
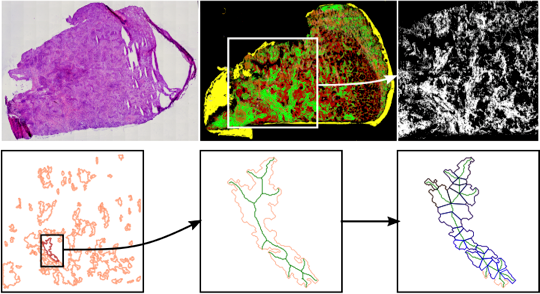
Die Laser-Capture Mikrodissektion (LMD) erlaubt es, aus Gewebedünschnitten bestimmte Bereiche hochpräzise auszuschneiden und molekular zu charakterisieren. Damit ist die LMD eine ideale Ergänzung zur in PRODI etablierten Infrarot-Mikroskopie. Um LMD im Kontext klinischer Studien optimal einsetzen zu können, müssen die zu extrahierenden Gewebereiche jedoch nach bestimmten geometrischen Kriterien in Teilformen zerlegt werden. Hierzu haben wir in Kooperation mit Prof. Maike Buchin (Fakultät für Informatik, RUB) entsprechende geometrische Optimierungsverfahren entwickelt.
Software-Plattformen für die Infrarot-Mikroskopie

In der Bioinformatik sind Software-Plattformen eine tragende Säule der wissenschaftlichen Infrastruktur. Offene wissenschaftliche Software gewährleisten Reproduzierbarkeit und ermöglichen Vergleichbarkeit unterschiedlicher Ansätze. So fördern Software-Plattformen die Etablierung neuer datengetriebener Technologien und beschleunigen den Transfer. Die methodischen Beiträge aus dem Kompetenzbereich Bioinformatik werden im Rahmen der DFG-geförderten OpenVibSpec Plattform verfügbar gemacht und tragen so zur Weiterentwicklung der Infrarot-Mikroskopie basierten Label-freien digitalen Pathologie bei.